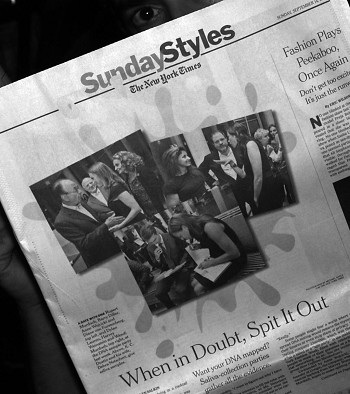
Imagínate despertar por la mañana, abrir tu correo electrónico y recibir el siguiente mail de la empresa que ha secuenciado tu genoma, lo tiene en su base de datos, y va actualizando la información que puedes extraer de él «Le informamos de que ayer la revista científica Nature publicó un estudio según el cual las personas con una mutación en el gen XYZ-987 tienen un 40 por ciento más de posibilidades de sufrir cáncer de colon que las personas sin esta mutación. Al contrastar con los datos que tenemos de su genoma, usted sí posee esa mutación. Ajustando con todo su perfil genético, su riesgo de padecer cáncer de colon ha aumentado a un 27 por ciento por encima de la media de la población. Le recomendamos que informe a su médico y adelante los controles de diagnóstico precoz a la edad que él establezca».
¿Ciencia ficción? En absoluto. Un escenario intermedio, que también parecía futurista hace diez años, ya se ha materializado.
Domingo, 14 de septiembre de 2008. Me había comprado el New York Times para sobrellevar las cuatro horas de autobús que me esperaban desde Nueva York a Washington D.C. Iba a ser un viaje tedioso y del que no esperaba emoción alguna, pero cuando al rato empecé a rastrear los diferentes suplementos del periódico, ¡sorpresa! En plena portada de la sección de ««Moda» del New York Times aparecía la foto de una elegante pareja escupiendo en un par de recipientes. La imagen ilustraba un reportaje sobre la recolecta de ADN acontecida el día anterior en un conocido local de la ciudad. No se trataba de una broma; la compañía de análisis genéticos 23andMe había organizado una fiesta para que miembros de la alta sociedad neoyorquina pudieran dejar muestras de su saliva en unos viales, devolverlos bien precintados a los responsables de la empresa, y recibir al cabo de un mes un informe genético personal con datos sobre el origen de sus antepasados, la predisposición a ciertas enfermedades comunes como el asma, la diabetes, la obesidad, o varios tipos de cánceres, y detalles como la predisposición a la calvicie, origen ancestral de sus antepasados, sensibilidad al sabor amargo, o saber si su cuerpo metabolizaba rápido la cafeína o no. En ese momento eran 89 los marcadores genéticos que estaba rastreando la compañía fundada por Linda Avey y Anne Wojcicki.
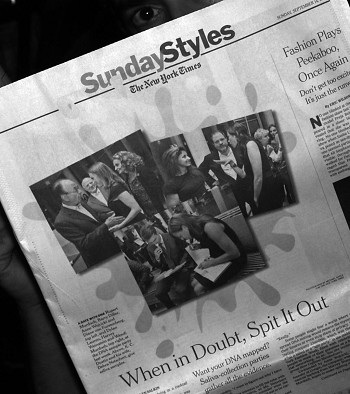
No exageremos. Ellas mismas reconocían que la información obtenida todavía no iba a ser demasiado relevante como diagnóstico médico. De ciertas enfermedades sólo analizaban alguno de los muchos factores genéticos que todavía faltaban por conocer, y sus clientes más bien debían considerarlo un divertimento o simple curiosidad. Sin embargo, algunos invitados parecían tomárselo más en serio, y el artículo recogía frases como: «Si mi marido tuviera un gen que hiciera peligrar seriamente a mi hijo en el parto quizá adoptaríamos», «Quiero averiguar por qué tengo pecas», o «En principio no dejaría a un novio aunque descubriera que no somos compatibles genéticamente, pero depende de lo que salga». Entre los asistentes había quienes ya se habían sometido con anterioridad a un test genético de la compañía e iban promulgando: ««Me hizo extremadamente feliz saber que tenía pocas posibilidades de padecer obesidad», o ««Me tranquilizó averiguar que un gen asociado a la enfermedad cardiovascular venía de mi madre, que tiene noventa y nueve años; porque mi padre murió a los sesenta y siete de un ataque cardíaco».
No consideres muy lejanas estas expresiones, porque dentro de muy poco tú mismo podrías estar repitiéndolas. De hecho, la tarifa de 23andMe por esos análisis era de unos muy asequibles 399 dólares, y pocos meses después, otras compañías ya estaban ofreciendo precios más competitivos y con mayor número de datos obtenidos a partir del ADN. No te extrañes si algún día alguien te regala por tu cumpleaños un test genético, o la posibilidad de secuenciar tu genoma completo.
Si algo reflejaba la fiesta del análisis de ADN, es que la tan ansiada era de la medicina personalizada estaba acercándose sigilosamente. Ese viaje en autobús me hizo recordar y constatar las palabras pronunciadas unos meses atrás por el prestigioso genetista Eric Lander en el MIT, cuando frente a un auditorio repleto dispuesto a escuchar la historia de su vinculación al Proyecto Genoma Humano, nos dijo: «El futuro ya ha llegado».
Cuando en junio de 2000 Francis Collins y Craig Venter anunciaron junto a Bill Clinton la primera secuenciación del genoma humano, advirtieron que en esos momentos su valor todavía era más simbólico que práctico. Los genetistas tenían en sus manos un libro extensísimo escrito en un código que cada vez conocían mejor, pero «ahora toca leerlo y encontrar el sentido de las frases», decían Ya se conocían bastantes mutaciones genéticas específicas que causaban por sí solas enfermedades como la fibrosis quística o la enfermedad de Huntington Pero caracterizar todas las variaciones relacionadas con trastornos que dependían de múltiples genes como la diabetes, los cánceres o las cardiopatías era una tarea lenta, muy lenta. Primero debían identificar genes asociados a características físicas o procesos metabólicos, luego ver si esos genes eran idénticos en todas las personas o si entre la población habían distribuidas versiones del mismo gen con pequeños cambios, y por fin comprobar si dichos cambios influían de manera significativa en su correcto funcionamiento. Llegar a caracterizar todos los genes relacionados con nuestras características físicas y poder establecer un riesgo genético fiable para enfermedades comunes parecía una empresa titánica. Todavía lo es. Pero como en muchas otras ocasiones en ciencia, la llegada de una herramienta tecnológica poderosísima ha acelerado exponencialmente el proceso.
Eric Lander fue así de entusiasta y contundente: «En 1997 se diseñó el primer chip para leer el ADN capaz de analizar si un gen específico tenía una mutación determinada o no. En 2001 apareció un nuevo chip que identificaba diez posibles mutaciones a la vez En 2002 ya conseguían analizar 1.000 posiciones; en 2004, 50.000; en 2006, 500.000, y en 2007 se llegó a un millón de polimorfismos genéticos individuales detectados en un único chip. ¡Esto, en manos de los investigadores, abre unas posibilidades de muestreo enormes!».
Con estos chips genéticos era mucho más sencillo agrupar personas con características diferentes, rastrear sus genomas de forma más exhaustiva, y comprobar si existía alguna mutación claramente asociada a un grupo de pacientes determinado. Los chips de ADN han multiplicado la capacidad de los científicos de interpretar la información genética, y los resultados no se han hecho esperar.
«Vayamos al grano —dijo Lander durante su presentación—. Olvidémonos de detalles superficiales como la altura, la apetencia por el sabor amargo, el color del pelo, o las enfermedades genéticas que sean minoritarias y ya sepamos identificar con otros métodos más específicos y directos. Hablemos directamente de cuántos polimorfismos genéticos individuales relacionados con enfermedades comunes como la diabetes, el infarto de miocardio, el glaucoma, el cáncer, se han descubierto en los últimos años». La tabla que nos mostró fue impresionante: en 2000 se identificó uno (asociado a la diabetes); en 2001, dos (de la enfermedad de Crohn); otro en 2002; uno en 2003; uno en 2004; cuatro en 2005; ocho en 2006; pero, de repente, entre enero y septiembre de 2007 se llevaban confirmados nada más y nada menos que 57 nuevos polimorfismos genéticos asociados a enfermedades comunes como cánceres de pecho, colon y próstata, diabetes I y II, obesidad, infarto de miocardio, artritis reumatoide, esclerosis múltiple, asma, y el ritmo se estaba acelerando exponencialmente. Todo eso gracias a los chips genéticos. El 2007 fue el annus mirabilis de la genética humana. El futuro había llegado. Una vez localizadas tantas mutaciones asociadas a patologías, por fin empezaba a tener sentido diseñar un nuevo tipo de chips para detectar fácilmente si uno las poseía o no. Y eso empezaron a hacer empresas privadas como 23andMe, DeCode, o Navigenetics.
Cautela era la palabra que repetían los genetistas más serios. Aunque, efectivamente, la progresión estaba siendo espectacular, todavía faltaba mucho genoma por leer y piezas que encajar para tener una visión sensata de nuestra salud a través del ADN. Además, los chips utilizados por las empresas privadas contaban con bastantes limitaciones. Una muy importante era que sólo medían mutaciones individuales o SNP (Single Nucleotide Polymorphisms), y hay muchas más formas en que un gen puede estar alterado y conducir a un mal funcionamiento. Pero, sobre todo, la restricción fundamental era —y continúa siéndolo— entender que la mayoría de nuestras características físicas o enfermedades habituales no dependen de un único gen, sino de muchos a la vez Conocemos algunas variaciones genéticas asociadas al infarto de miocardio, pero seguro que existen muchas más no descubiertas todavía. La lectura del genoma será un proceso continuo que probablemente no terminará nunca, y esto nos conduce a la visión futurista con que abría este texto. Secuenciar el primer genoma humano completo costó 3.000 millones de dólares; el de James Watson «sólo» un millón; Craig Venter secuenció de nuevo el suyo por 300.000 dólares; y se convocó el Premio X de 10 millones de dólares al primer equipo que fuera capaz de secuenciar cien genomas humanos en diez días por menos de 10.000 dólares cada uno. Visitando un laboratorio del MIT en mayo de 2008, me mostraron el secuenciador que les había llegado la semana anterior diciendo: «Hace en dos semanas lo que todo el Proyecto Genoma Humano hizo en tres años y con un equipo de centenares de investigadores». Quién sabe cuánto costará secuenciar tu genoma cuando leas estas líneas, pero sin duda podrás permitírtelo. En junio de 2010 tuve la oportunidad de entrevistar personalmente a Francis Collins con motivo del décimo aniversario de la presentación del primer borrador del genoma humano. La frase que más me impactó fue: «Me sorprendería mucho que dentro de diez años, tú y yo —o cualquiera que tenga acceso al sistema sanitario— no tuviéramos nuestro genoma secuenciado. Los beneficios de conocer esta información serán enormes, y el coste se está desplomando». Quedan grandes retos científicos, técnicos, sociales y éticos que afrontar para poder sacar el máximo partido a dicha información. Pero, sin duda, se acerca el día en que el genoma formará parte de nuestro historial médico, estará informatizado, y se irá actualizando a medida que los científicos vayan descubriendo más asociaciones a enfermedades, características físicas, habilidades mentales o incluso predisposición a ciertos comportamientos: «Su colesterol es de 278, la presión sanguínea ligeramente alta, sobrepeso, no fumador, nivel de azúcar correcto, predisposición genética 62 por ciento por encima de la media», podría decirte tu médico.
Respecto a la predisposición genética y la prevención, tampoco tengas grandes expectativas. Salvo pocos errores fatales que condicionan fuertemente a ciertas enfermedades, como el BRCA 1 y 2 para el cáncer de mama y otros, la suerte genética quedará repartida. Me refiero a que los científicos están descubriendo tantísimos genes implicados en características comunes que en raras ocasiones la lotería genética te habrá castigado con todas las mutaciones dañinas, o premiado con una protección excepcional para evitarlas. En realidad, todos somos muy iguales, de la misma manera que poca gente sobrepasa los dos metros de altura o se queda en el metro cincuenta. Conocerás en mayor detalle tus riesgos, qué fármacos son los mejores para ti, y a qué aspectos de tu salud debes prestarles más atención. Pero en casos normales, el dato genético podrá quedarse en una simple línea más, y tu destino será decidido por cómo trates a tu cuerpo o elijas utilizar tu cerebro.
Otra historia es cuando tu organismo empiece a funcionar mal. Ahí, sin embargo, la información genética sí podría salvarte la vida. Veamos un ejemplo concreto de esto último, que no es tremendamente relevante, pero sí muy ilustrativo: en enero de 2010 científicos de los Institutos Nacionales de la Salud descubrieron que uno de los cánceres cerebrales más comunes, el glioblastoma, podía estar causado por cuatro mutaciones genéticas diferentes. Hasta el momento, como los tumores del glioblastoma siempre tenían la misma apariencia clínica, los médicos lo habían considerado un único tipo de cáncer. Pero ahora veían que a nivel molecular, había cuatro subtipos. Y lo más importante, cada subtipo reaccionaba de manera diferente a la quimioterapia y otros tratamientos. Es ahí donde aparece la revolución. Las terapias específicas no son nuevas en absoluto, pero la dimensión que está adquiriendo conocer a fondo el perfil genético de ciertas enfermedades, especialmente el cáncer, hace que por fin podamos asegurar que la expansión de la tan ansiada medicina personalizada está a la vuelta de la esquina. O «ya estamos inmersos en ella», como asegura Francis Collins al recordarnos que la implantación no llegará de un día para otro, sino que será un progresivo goteo de conocimientos y aplicaciones concretas que cambiarán poco a poco la manera de afrontar enfermedades.
«Tómese este fármaco específico y en esta cantidad ajustada a su perfil genético», te dirá el médico dentro de muy poco tiempo. O «Este producto para la calvicie es el que mejor funciona para una mutación como la tuya», te aconsejará el esteticista genético.
Insisto en lo de muy poco tiempo. Tener tu genoma secuenciado será rutinario en menos de una década. No puede ser de otra manera. El precio de hacerlo se está reduciendo a un ritmo impensable hace unos pocos años, inversamente proporcional a la cantidad de valiosísima información que de él podemos obtener. Los progresos en investigación básica están siendo incluso más rápidos de lo que imaginaban los científicos. Ahora falta el dificilísimo reto de trasladarlo a la práctica médica, pero el punto de inflexión ha llegado: ya hay suficiente masa crítica de conocimiento en genética humana para plantearse aplicarlo a gran escala, e integrarlo definitivamente al cuidado cotidiano de nuestra salud.
Una medicina preventiva específica y adaptar fármacos a nuestros genes estropeados será el primer paso. No está tan cerca, pero cada vez menos lejos, el momento en que podamos llegar a repararlos y evitar transmitirlos a nuestros hijos.