4
Margulis no basta
La teoría de la endosimbiosis serial de Margulis es obviamente mucho más interesante, sensata y realista que los dogmáticos balbuceos darwinistas para explicar el origen de la célula eucariota. Pero también es obviamente incompleta. En este capítulo expondré por qué, basándome en tres desconcertantes misterios revelados por la biología molecular.
Misterio 1. El splicing
Para leer esta frase, usted ha empezado por la izquierda, está ahora mismo progresando hacia la derecha y dentro de poco alcanzará el punto final. Al hacerlo, usted no ha encontrado frases intercaladas del tipo «No siga usted leyendo esto, por Dios» o «Quite esta frase de aquí y el resto tendrá sentido». Los seres vivos, sin embargo, se encuentran muy a menudo con ese tipo de estorbos cuando leen sus genes. Se llaman intrones. Son segmentos de ADN que aparentemente no hacen nada útil y están colocados, de manera incomprensible, en mitad de los genes. Después de leerlos, la célula debe detectarlos, desgajarlos de la información propiamente dicha, y volver a pegar los trozos restantes afinando exactamente a la letra, con extrema precisión. Este costoso proceso recibe el eufónico nombre inglés de splicing (que viene a querer decir «empalmar»), pero se trata en realidad de un verdadero engorro. Una rémora que comparten, inexplicablemente, todos los animales y plantas, y gran parte de los protistas: es decir, la generalidad de los eucariotas.
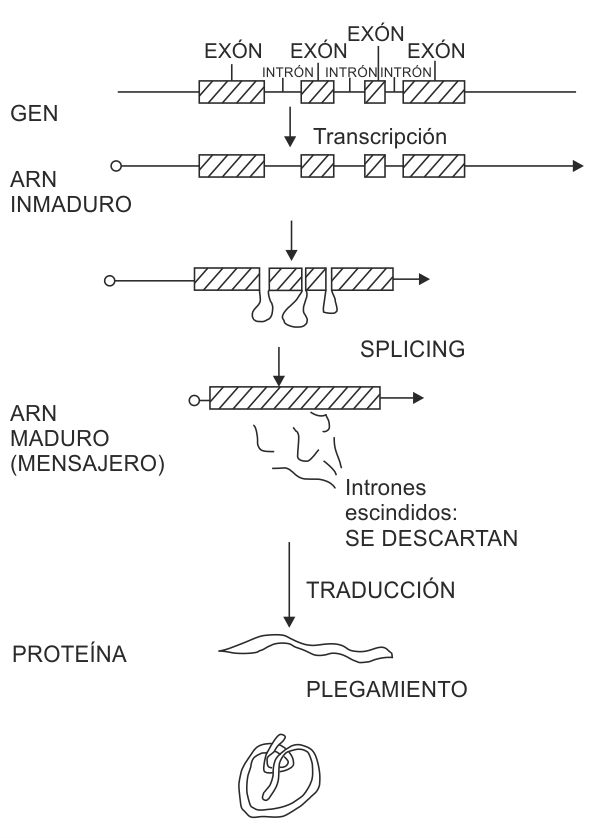
FIGURA 4.1: La información de los genes está interrumpida por segmentos de ADN sin sentido, llamados intrones. Para poder leer los genes correctamente, la célula debe primero eliminar los intrones del ARN mensajero, en el proceso conocido como splicing.
¿Qué diseñador, qué especie de ingeniero, qué clase de Creador hubiera dispuesto las cosas de una manera tan estúpida? ¿A qué vienen, señor diseñador, esos malditos intrones? ¿Quién los encargó? ¿No será mejor leer los genes de un tirón, como se lee cualquier texto, cualquier melodía tonal o dodecafónica, cualquier secuencia cinematográfica o cualquier programa informático inventado por el ser humano? ¿Para qué demonios esas interrupciones absurdas que luego hay que tomarse el fastidoso trabajo de quitar de en medio? ¿Qué diría ahora el reverendo Paley, máximo exponente del argumento teológico del diseño que pretendía demostrar la existencia de Dios a través de la perfección de sus obras biológicas?
Pues, por mentira que parezca, nos podemos hacer una idea bastante cabal de lo que hubiera dicho Paley. Habría dicho: «Los intrones están ahí porque permiten a las especies evolucionar más eficazmente». ¿Cómo? ¿Paley hablando de evolución? ¿Hasta dónde va a llegar esta farsa?
Pues sí. Paley hubiera tenido que hablar de evolución porque no le hubiera quedado otro remedio. En los casi 30 años de exhaustiva investigación que han pasado desde que se descubrió el splicing, nadie ha podido intuir qué utilidad, por mínima y retorcida que sea, pueden tener los desconcertantes intrones para la célula (o el organismo) que los alberga. Con una excepción: la utilidad evolutiva. La idea, que fue propuesta en los años setenta por William F. Doolittle y Walter Gilbert, consiste en lo siguiente: si los genes están divididos en segmentos (llamados «exones» por oposición a los «intrones» que los interrumpen), será porque cada segmento (cada exón) es, de alguna forma, una unidad funcional. No una unidad funcional del gen, porque el gen no es más que un texto, y tener que dividirse en segmentos es un verdadero estorbo para él, como para cualquier texto. Pero sí una unidad funcional de la proteína que el gen significa (véase glosario). Muchísimas proteínas están, en efecto, hechas de segmentos especializados: uno para catalizar una reacción química, otro para alimentarla de energía, otro para interactuar con otra proteína, otro para anclarse a cierta membrana de la célula, o para colarse en uno u otro de sus compartimentos. ¿No será que cada exón, es decir, cada segmento de gen comprendido entre dos intrones, corresponde a un segmento especializado de la proteína? Se trata de una idea simple, y fácil de someter a prueba. El resultado, por lo que sabemos hoy, es que algo hay. Algunos exones, aunque desde luego no todos, corresponden a segmentos especializados de la proteína. No es perfecto, pero casi nada en biología lo es: tal vez la correspondencia fue estricta en el pasado, y la evolución posterior lo ha complicado todo. Demos una oportunidad a Doolittle y Gilbert, y veamos adonde nos lleva su idea.
Si los exones —algunos de ellos, al menos— se corresponden con los segmentos funcionales de las proteínas, ¿qué sacaríamos con ello, si los genes no se aprovechan ni lo más mínimo de esa armoniosa correlación semántica? Los genes pueden estar todo lo divididos en exones que quieran, pero lo primero que hacen antes de dar a leer su información es librarse de las interrupciones y pegar todos los exones juntos en el mismo orden en el que ya estaban. Es decir: dejar las cosas como siempre hubieran sido de no existir los intrones. La célula no se entera en ningún momento de que la información del gen estaba inicialmente dividida en esos paquetes semánticos tan elegantes. Si no lo hubiera estado, el resultado hubiera sido el mismo: una sola proteína, un solo texto lineal, compuesto de varios segmentos funcionales, sí, pero sin interrupción alguna entre uno y otro. ¿Entonces qué sacamos de todo esto?
Sólo una cosa: que los genes podrían evolucionar más eficazmente, y por lo tanto hacer evolucionar a las especies más rápidamente. Ya se lo dije al lector: la única salida de Paley era la evolución. Y ésa es precisamente la razón de que Doolittle y Gilbert formularan la teoría de los exones y los segmentos especializados. ¿Qué mejor forma de evolucionar que barajando de vez en cuando los exones, obteniendo así nuevas proteínas, nuevas combinaciones de viejos módulos, nuevos dispositivos biológicos formados sin necesidad de empezar desde cero todo el trabajo de diseño? Este barajado de los genes en la evolución se vería enormemente facilitado por la división de los genes en exones, ya que las nuevas junturas no tendrían por qué darse en un lugar muy preciso. Cualquier unión improvisada chapuceramente en cualquier punto del intrón serviría. La maquinaria del splicing se encargaría luego de eliminar el chapucero intrón y pegar los dos exones, vecinos en un gen por primera vez en la historia, exactamente por el punto en que se pegaban antes a unos vecinos distintos. Sin nefastas incongruencias en la pauta de lectura. Sin añadir o quitar elementos entre los dos nuevos textos vecinos (figura 4.2).
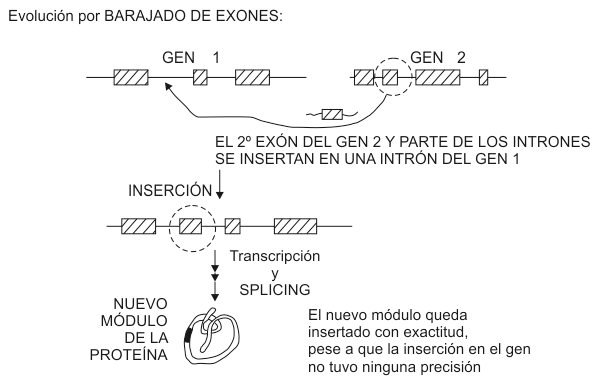
FIGURA 4.2: El hecho de que los genes contengan intrones plantea grandes ventajas para la evolución de nuevas proteínas. La incorporación de un nuevo módulo a una proteína puede basarse en un suceso muy impreciso. El splicing hace luego el resto.
Éste es el ejemplo más consumado que conozco de lo que podríamos llamar el argumento metadarwinista. Sí, he dicho meta: no es ya que las cosas sean como son porque eso las hace un poco más eficientes que sus predecesoras, como nos diría Darwin, sino que las cosas son como son porque eso les puede reportar en el futuro lejano la gran ventaja de evolucionar darwinianamente hacia otra cosa impredecible. La verdad es que el argumento se las trae. El metadarwinismo, posiblemente, habría repugnado profundamente a Darwin, que hizo enormes esfuerzos intelectuales por mantenerse pegado a una teoría lo más honrada posible, basada en mecanismos estrictamente locales, transitorios, ciegos, carentes de previsión, de sentido, de estrategia. La evolucionabilidad no es una parte natural de la teoría de la evolución de Darwin. Y de hecho, la ortodoxia darwinista siempre se ha sentido muy incómoda con ese concepto.
Pero eso no invalida el metadarwinismo como posibilidad teórica. Los argumentos a favor del metadarwinismo pueden llegar a ser un poco rebuscados, pero son lógicamente admisibles, y vienen a sonar como sigue. De las especies existentes en cualquier momento, todas igualmente eficaces y adaptadas, siempre habrá algunas que, por mero azar, sean más flexibles, más adaptables, más evolucionables que otras. Ese mero hecho no les sirve de nada en principio, desde luego. Pero cuando sobreviene una crisis —un cambio drástico del entorno, la irrupción de un nuevo predador, el ya clásico meteorito—, las especies más rígidas lo pasan muy mal para subsistir, mientras que las más evolucionables pueden salvarse por el simple prospecto de, bueno, pues eso, de evolucionar hacia otra cosa. Y ahora viene lo más importante: A lo largo de millones o de miles de millones de años, habrán tenido lugar tantas crisis que la evolucionabilidad habrá acabado por evolucionar, por hacerse cada vez más sutil, eficaz y previsora. De ahí los intrones.
Los argumentos metadarwinistas, como se ve, suelen estar en el límite de la desintegración del darwinismo. Pero no son absurdos. Por muy útil que acabara resultando en el futuro, nada impide que el splicing pudiera surgir como una chapuza histórica de la que es muy difícil librarse. Al fin y al cabo, el splicing, por complicado que parezca, podría basarse simplemente en una o dos proteínas capaces de cortar a los ácidos nucleicos en ciertas posiciones, y de volverlos a pegar en ciertas otras, y éstas son funciones que seguramente serían necesarias de todos modos, aunque el splicing no hubiera hecho nunca su ridícula aparición. El splicing pudiera muy bien ser un mero efecto secundario de invenciones evolutivas más básicas y mucho más importantes en el corto plazo: el único plazo que Darwin nos permite utilizar como argumento evolutivo. Su gran utilidad para la evolucionabilidad de su poseedor sería un efecto colateral, que sólo se dejaría sentir mucho más tarde. Ésta es la justificación de los argumentos metadarwinistas, en éste y otros casos.
Pero no funciona, al menos en este caso. El dispositivo molecular que se ocupa de eliminar los intrones del ARN mensajero y pegar juntos los exones para permitir una lectura de corrido, una lectura que quiera decir algo, recibe el horrible nombre de spliceosoma, y comprende cerca de un centenar de proteínas y media docena de pequeñas moléculas de ARN. El spliceosoma es muy parecido en todos los eucariotas, y es obvio por ello que debió inventarse antes de que los protistas, los hongos, los animales y las plantas emprendieran sus caminos evolutivos en solitario. Es decir, hace unos mil o dos mil millones de años. Pero si el splicing se basa en un mecanismo tan complicado, ¿de dónde diablos lo sacaron los eucariotas?
Si la teoría de Margulis es correcta, el splicing debe provenir de alguno de los procariotas que se asociaron simbióticamente para crear la primera célula eucariota. Pero ningún procariota conocido tiene nada parecido a un spliceosoma. Bien, intentemos simplificar las cosas. Pese a la exuberante diversidad de proteínas que contiene el spliceosoma, sus componentes esenciales son los pequeños ARNs, que son los que hacen directamente el trabajo de eliminación de los intrones. Para entender el origen del splicing, por lo tanto, es imprescindible saber de dónde salieron estos pequeños ARNs. ¿Hay en las bacterias algo parecido a los pequeños ARNs del spliceosoma eucariota?
Las bacterias no tienen nada parecido a un spliceosoma, pero sí tienen intrones en algunos de sus genes, y tienen por tanto que deshacerse de aquéllos para poder leer éstos. Y, curiosamente, también usan una molécula de ARN para catalizar la reacción de escisión de los intrones. Lo que ocurre es que ese ARN es… ¡el propio ARN del intrón! Estos intrones bacterianos, que se conocen como «intrones del grupo II», poseen en sí mismos la actividad enzimática necesaria para escindirse del ARN mensajero. Y ahora viene lo más importante: las reacciones químicas implicadas en esa escisión son exactamente las mismas que las realizadas por el spliceosoma en los organismos eucariotas. Es posible, por tanto, que los pequeños ARNs del spliceosoma se originaran en la evolución como fragmentos de un intrón bacteriano del grupo II. Nada de esto se opone a la teoría de Margulis, siempre que uno suponga que el centenar de proteínas del spliceosoma no sirve en realidad de gran cosa, y que, por así decir, se apuntaron a ese bombardeo en los inicios de la evolución eucariota por la sencilla razón de que nada se lo impidió en aquellos tiempos difíciles.
Incluso después de todas estas concesiones, sin embargo, queda un problema bastante grave. En los organismos eucariotas, el splicing es sólo un paso del intrincado proceso que se ocupa de leer la información contenida en los genes. (El término técnico para esta lectura es expresión genética). La versión de libro de texto de este mecanismo sigue siendo el esquema simple formulado en los años sesenta por Crick, Watson, Brenner, Jacob y Monod: el ADN que forma un gen se transcribe (es decir, se copia) en una molécula similar llamada ARN mensajero, que tiene la misma secuencia de bases. Mientras el ADN permanece en el núcleo, el ARN mensajero sale al citoplasma de la célula, donde su texto escrito en bases se traduce en una secuencia precisa de aminoácidos, es decir, en una proteína. Este modelo básico sigue siendo válido en lo esencial cuarenta años después de su formulación, pero los detalles han resultado ser horrorosamente complicados.
Hoy sabemos que el proceso de lectura (expresión) de los genes requiere varias máquinas multiproteicas (es decir, compuestas por varias proteínas) exquisitamente acopladas en todos los eucariotas, desde las miserables amebas unicelulares hasta los gloriosos seres humanos. Una máquina multiproteica se ocupa de iniciar la transcripción del ADN en ARN; otra de elongar la naciente molécula de ARN, y aún otra de finalizar esa elongación; en paralelo, y nada más comenzar el recién mencionado proceso de transcripción, otras máquinas multiproteicas sellan el extremo inicial del ARN, realizan el splicing y añaden una etiqueta (consistente en una ristra de letras A) en el extremo final. Otras máquinas multiproteicas exportan el ARN, ya liberado de sus intrones y bien acicalado en sus dos extremos, desde el núcleo hasta el citoplasma, donde será traducido en una proteína. Esta traducción, por cierto, se lleva a cabo en otra máquina llamada ribosoma. En los últimos años ha ido resultando más y más evidente que estas máquinas multiproteicas no son independientes: la que inicia la transcripción interactúa (y comparte elementos) con la que se ocupa de la elongación, que a su vez mantiene contactos con la que la termina, etcétera. Pero los datos más recientes revelan un grado de integración que va mucho más allá de esos contactos más o menos vecinales. Todas las máquinas del proceso están profundamente interconectadas. Por ejemplo, la primera, que inicia la transcripción, comparte elementos con la última, que exporta el ARN maduro al citoplasma. En una reciente revisión de este problema, los científicos de Harvard Tom Maniatis y Robin Reed concluyen:
La vasta red de acoplamientos discutida aquí revela que virtualmente todos los pasos de la expresión genética [la lectura de los genes], desde el primero hasta el último, están acoplados. La imagen que emerge es que la expresión genética se lleva a cabo en una factoría inmovilizada, consistente en un gran número de máquinas interactivas, que orquesta los múltiples pasos del proceso. (MANIATIS y REED, 2002.)
Éste es el problema grave al que me refería, porque la máquina del splicing (el spliceosoma) conforma el mismísimo centro de esa factoría, y establece una complejísima, y exquisitamente ajustada, red de interacciones con todas las demás máquinas multiproteicas de la factoría: con las máquinas de iniciación, elongación y terminación de la transcripción de ADN en ARN, con las que sellan el extremo inicial del ARN y etiquetan su extremo final, y con las que exportan el mensajero desde el núcleo hasta el citoplasma. El splicing no parece provenir ni de una molesta chapuza añadida secundariamente al esencial dispositivo de la transcripción, ni de un inevitable accidente al que la evolución encontró después la utilidad de la evolucionabilidad: el splicing está integrado hasta el cuello en el mismísimo centro lógico de la factoría para leer genes que utilizan todas las especies de protistas, hongos, plantas y animales, seguramente desde la mismísima invención de la célula eucariota.
El splicing dota con toda probabilidad a los organismos que lo poseen de una notable creatividad y evolucionabilidad, gracias a que ofrece una forma fácil de barajar los exones de los genes y crear así nuevas proteínas. Pero su aparición no parece responder a un progresivo incremento de la evolucionabilidad, forjado a base de extinciones masivas que barrieron del mapa a todas las especies excepto a las que, como efecto secundario e impredecible de algún otro mecanismo, tuvieron la suerte de poseer una mayor flexibilidad evolutiva. Al menos en este caso, la evolucionabilidad es una propiedad de un sistema complejo, inventado una sola vez en la historia, sin formas transitorias obvias, integrado en el mismísimo epicentro de una factoría esencial del núcleo eucariota, y que ha permanecido esencialmente íntegro desde su aparición, hace tal vez unos mil millones de años. La evolucionabilidad nunca cuadra muy bien con el darwinismo, pero es que en este caso ni siquiera parece cuadrar con el metadarwinismo. Y la factoría como tal no existe en las arqueas ni en las bacterias, con lo que la teoría de Margulis también se ve en dificultades en este caso. ¿Por dónde se sale de este túnel? De momento, por otro túnel aún más oscuro: el cronocito. Siga leyendo.
Misterio 2. El cronocito
Si lo que venimos denominando el cuerpo central de la célula eucariota, es decir, la estructura básica que todavía carece de mitocondrias y cloroplastos, se formó mediante la simbiosis de una arquea y una bacteria, como propone la teoría de Margulis, el genoma eucariota fundamental —el conjunto de genes compartidos por protistas, hongos, plantas y animales— debería consistir en la suma de los genomas de la arquea y la bacteria que intervinieron en la fusión. Naturalmente, muchos de esos genes serían redundantes (la arquea y la bacteria debían tener cientos o miles de genes muy parecidos) y podrían resultar eliminados más tarde. Y cada reino eucariota —protistas, hongos, plantas y animales— habrá sido muy libre después de incorporar nuevos genes específicos y peculiares. Pero los genes compartidos por los cuatro reinos eucariotas debían estar ya presentes en el ancestro común de todos ellos, y cabría esperar que hubieran sido aportados bien por la arquea, bien por la bacteria. Esta predicción se cumple para muchos genes —por ejemplo para los analizados por Radhey Gupta, que vimos en el capítulo anterior—, pero no para todos.
Hyman Hartman, del Massachusetts Institute of Technology (MIT), y Alexei Fedorov, de la Universidad de Harvard, han aprovechado los genomas ya conocidos para poner a prueba esa predicción. Primero han buscado el genoma eucariota fundamental: aquellos genes compartidos por Giardia lamblia (un protista), la levadura de la cerveza (un hongo), la mala hierba Arabidopsis thaliana (una planta) y dos animales: el gusano Caenorhabditis elegans y la mosca Drosophila melanogasier. Según sus datos, ese genoma fundamental eucariota está compuesto por 2.136 genes. (HARTMAN y FEDOROV, 2002.)
A continuación, Hartman y Fedorov se han preguntado cuántos de esos genes provienen de la bacteria y de la arquea que intervinieron en la fusión. No han hecho ninguna suposición sobre qué bacteria y qué arquea concretas podían ser: se han limitado a preguntarse cuáles de los 2.136 genes del genoma eucariota fundamental pueden encontrarse en cualquier bacteria o arquea de la cincuentena de especies cuyos genomas se han secuenciado ya. La mayoría de esos genes (1.789) existen en bacterias o en arqueas, y por lo tanto pudieron muy bien ser aportados por éstas durante el suceso simbiótico. Pero los otros 347 genes no tienen equivalentes en ninguna bacteria ni en ninguna arquea conocida. ¿De dónde salieron, entonces? He aquí el misterio.
En realidad, el misterio es mucho más profundo de lo que sugiere esa simple cifra de 347 genes. El verdadero enigma aparece cuando averiguamos de qué se ocupan esos genes. Vimos en el capítulo pasado que, en líneas generales, la bacteria (fuera o no una espiroqueta) había aportado a la fusión los genes del metabolismo, la cocina química de la célula, y que la arquea había aportado los genes autoalusivos que se ocupan de procesar y manipular la propia información genética: replicar el ADN, transcribirlo en ARN y traducirlo al lenguaje de las proteínas. ¿A qué se dedican los famosos 347 genes que no provienen ni de las bacterias ni de las arqueas? Les advierto que la respuesta es muy extraña, por si prefieren no seguir leyendo.
Sabemos que el paso evolutivo de los procariotas (arqueas y bacterias) a los eucariotas es la mayor discontinuidad de la historia de la Tierra. La diferencia más vistosa entre ambos tipos de células es la que da nombre a ambos grupos: los eucariotas tienen su genoma empaquetado en un núcleo, y los procariotas no tienen núcleo. Pero hay otros tres dispositivos o procesos, esenciales y altamente complejos, que poseen todos los eucariotas y no posee ningún procariota:
1. La endocitosis, un sistema muy organizado que sirve para engullir a otras células, o fragmentos de células, o virus o moléculas grandes, y para degradarlos en condiciones controladas dentro de unas vesículas (esferitas membranosas) que se fusionan, se separan y se trasladan por el interior de la célula en una coreografía complicada y precisa.
2. El sistema de transducción de señales, una maraña inextricable de proteínas que modifican a otras proteínas que modifican a otras proteínas, utilizada por todas las células eucariotas para comunicar (transducir) los mensajes que reciben del exterior (hormonas, contactos con otras células, factores que estimulan la división celular, etcétera) a los centros logísticos del interior de la célula.
3. La factoría del núcleo. El hecho de tener el genoma empaquetado en un núcleo conlleva la necesidad de disponer de un sistema de puertas (poros nucleares) que den paso de forma selectiva y regulada a los componentes celulares que se fabrican en el citoplasma y deben luego entrar al núcleo (centenares de proteínas) y a los que se fabrican en el núcleo y deben salir al citoplasma (como los ARNs mensajeros). Estas máquinas de tráfico aduanero están asociadas a varias otras máquinas que condensan o relajan con precisión a las distintas partes del material genético, y que se ocupan de su activación modulada, de la regulación de su transcripción y otros procesos nucleares exquisitamente acoplados. Uno de ellos es el splicing, que vimos en detalle en la sección anterior.
Éstas son las tres marcas de fábrica de los eucariotas: los tres dispositivos complejos que todos los eucariotas comparten y que ningún procariota posee. Si la primera célula eucariota evolucionó por la simbiosis de dos (o más) células procariotas, cabría conjeturar que esos tres dispositivos complejos surgieron de la suma de partes más simples aportadas por los procariotas que intervinieron en la fusión, por más que el sistema sufriera con posterioridad toda clase de complicaciones y ajustes. Pero los análisis comparativos de Hartman y Fedorov parecen fulminar esa hipótesis. Porque los genes necesarios para construir las tres marcas de fábrica eucariotas (la endocitosis, el sistema de transducción de señales y la factoría del núcleo) no parecen provenir ni de la bacteria ni de la arquea que intervinieron en la fusión: ¡Son precisamente los famosos 347 genes que comparten todos los eucariotas y que no están presentes en ningún procariota conocido! Para ser más exactos, de esos 347 genes exclusivos de los eucariotas, 91 están relacionados con la endocitosis, 108 con la transducción de señales y 47 con las máquinas del núcleo (la función de los 101 restantes se desconoce por el momento). ¿Qué demonios pasa aquí?
Vaya por delante que estos hechos no refutan la teoría de Margulis: el 85% del genoma eucariota fundamental está constituido por genes de bacterias y de arqueas, y ello viene en apoyo de la teoría simbiótica de Margulis. Pero que el otro 15% del genoma eucariota fundamental, precisamente los 347 genes que no existen ni en arqueas ni en bacterias, sea precisamente el responsable de las tres marcas de fábrica del mundo eucariota no es la clase de cabo suelto con la que uno puede dormir tranquilo. Si la célula eucariota se formó por simbiosis, ¿cómo se traga que la esencia de la eucariotez haya conseguido escapar tan nítidamente del mecanismo simbiótico que dio origen a la mismísima eucariotez? No tiene sentido, y para mí es obvio que se nos está escapando algo muy fundamental.
La solución propuesta por Hartman y Fedorov es acudir a un tercer hombre. La célula eucariota (esto es, su cuerpo central) no se formó por la fusión de dos microbios, sino de tres: una bacteria, una arquea y lo que ellos llaman un cronocito: un tercer microorganismo que aportó los 347 genes responsables de la eucariotez. Es indudable que esta hipótesis resuelve matemáticamente la paradoja. Pero también es verdad que parece muy traída por los pelos. Las bacterias y las arqueas han estado siempre y siguen estando por todas partes, pero del tal cronocito nadie tiene la menor noticia. Más aún: si el cronocito era uno de los microbios que construyeron por simbiosis a la primera célula eucariota, es obvio que no podía ser un eucariota. Y si era un procariota, ¿para qué quería la endocitosis, el sistema de transducción de señales y, sobre todo, la factoría del núcleo? Demasiadas preguntas, y demasiado difíciles de responder.
Y, sin embargo, hay muchas más especies de bacterias de las que conocemos, e incluso algunas de las que conocemos parecen escondernos sus mejores cartas. Déjenme llamar su atención sobre una de ellas, llamada Pirellula. Es una bacteria, pero carece de las paredes celulares típicas de las bacterias. Maneja un grupo de grasas —ácidos palmítico, oleico y palmitoleico— que parecen más propias de los eucariotas que de las bacterias. Se multiplica por gemación, un mecanismo típico de eucariotas como los hongos y desconocido en otras bacterias. Y, colmo de los colmos, guarda su ADN en una estructura superficialmente parecida a un núcleo. Según los más recientes árboles genealógicos, Pirelulla y sus primas, una familia de bacterias denominadas planctomicetos, son el grupo de microorganismos procariotas más antiguo de la Tierra (Brochier y Philippe, 2002.) Nadie sabe aún si Pirellula puede tener algo que ver con el hipotético cronocito de Hartman y Fedorov, pero mientras escribo este capítulo (mayo de 2002) varios laboratorios del mundo se están apresurando a secuenciar el genoma de estos extravagantes microbios. Parece una buena idea, desde luego.
Misterio 3. Las máquinas
Las máquinas exquisitamente acopladas no son una peculiaridad del sistema de lectura o expresión genética que ya hemos visto dos veces en este capítulo. En enero de 2002, un equipo de 38 investigadores de la empresa Cellzome —una compañía fundada enteramente por científicos del Laboratorio Europeo de Biología Molecular (EMBL), en Heidelberg (Alemania)— presentó en Nature los resultados de la primera búsqueda sistemática de máquinas multiproteicas. Este equipo, dirigido por el italiano Giulio Superti-Furga, analizó de un golpe unos 1.400 genes (una tercera parte del genoma) de la levadura Saccharomyces cerevisiae, el hongo unicelular que los panaderos utilizan para hacer el pan, los cerveceros para hacer la cerveza, y los genetistas para hacer la pascua a sus colegas que estudian a otras especies eucariotas muchísimo más lentas y difíciles de manipular, como la mosca o el ser humano. El resultado fue una de las mayores sorpresas científicas de los últimos años: las 1.400 proteínas fabricadas por esos 1.400 genes no vagan en solitario por la célula, cada una aportando su pequeña cuota de know how a la empresa celular, sino que todas están formando parte de máquinas multiproteicas. Para ser exactos, las 1.400 proteínas analizadas por el equipo de Heidelberg constituyen 232 máquinas. La máquina más pequeña está formada por sólo dos proteínas. La más grande, por 83. Una máquina media está compuesta por 12 proteínas (no se moleste en dividir 1.400 por 232: no da 12,sino 6; veremos la razón dentro de dos párrafos).
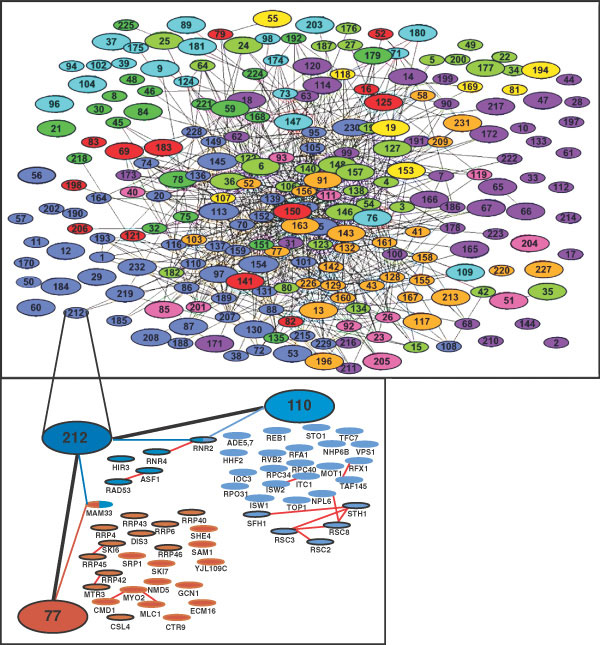
FIGURA 4.3: Malos tiempos para quienes creían que las proteínas andan Dolando por la sopa celular sin orden ni concierto. En el recuadro de abajo, cada elipse pequeña representa una proteína. Se ven dos constelaciones, y cada una representa una máquina (elipses grandes). Arriba se representan las 242 máquinas proteicas descubiertas en Heidelberg: cada línea representa una interacción estable entre dos máquinas (elipses). La maraña es inextricable porque casi todas las proteínas forman parte de máquinas complejas, que a su vez forman parte de grandes factorías celulares.
¿A qué se dedican estas máquinas? Según el amplio muestreo del equipo de Heidelberg, la mitad de las máquinas están implicadas en la manipulación y utilización del material genético: transcripción de los genes a ARN y estructura de la cromatina (24%),splicing y metabolismo del ARN (12%) y síntesis y retirada de proteínas (14%). (Subtotal: 50%). Otro 19% de las máquinas están dedicadas al metabolismo energético. Otro 9% a construir las membranas de la célula. Otro 9% a transmitir señales. Un 6% al ciclo celular que ordena el crecimiento y la división, y un 3% a dotar a la célula de estructura y polaridad. (GAVIN y colaboradores, 2002.)
Pero las redes de interacciones no se limitan a los componentes de una máquina. Hay numerosas proteínas que pertenecen a varias máquinas a la vez (por eso no le hubiera salido la división). En algunas ocasiones, esto refleja una interacción estable entre varias máquinas. Otras veces, esa interacción es sólo transitoria, dependiente de la fase del ciclo celular, las condiciones del entorno u otros factores. Cuando los científicos de Heidelberg representaron en un solo mapa todas estas interacciones, descubrieron un hecho inesperado más: la gran mayoría de las máquinas están asociadas entre sí, directa o indirectamente, a través de proteínas comunes (figura 4.3.) «En una primera aproximación, toda la célula es una sola máquina», en palabras de Cayetano González, investigador del EMBL.
A nadie sorprenderá a estas alturas saber que estas máquinas muestran un altísimo grado de universalidad en todas las células eucariotas. Los estudios en humanos son todavía muy preliminares, pero la mayoría de las máquinas identificadas en levaduras existen también en nuestra especie. No es que una proteína o dos estén conservadas entre la levadura y el ser humano: es que la máquina en su conjunto es esencialmente la misma en todas las especies eucariotas, aunque sólo la mitad (la cifra varía) de las proteínas individuales que la componen son las mismas. La evolución ha jugado con estas máquinas de alta precisión sustituyendo algunos componentes, pero respetando su estructura básica y la lógica de su funcionamiento. Según Luis Serrano, otro científico del laboratorio europeo de Heidelberg, cada proteína está implicada en tantas interacciones con las otras proteínas de la máquina, o con las proteínas de otras máquinas, que prácticamente no queda sitio disponible en la superficie de la proteína para añadir nuevos componentes. Pero siempre será posible sustituir un componente por otro.
La organización de la célula en máquinas altísimamente estructuradas supone una enorme restricción a los mecanismos evolutivos concebibles. En palabras de Superti-Furga: «Las levaduras y las células humanas comparten un altísimo número de máquinas similares, compuestas por proteínas relacionadas. Esto significa que, mientras cada proteína ha cambiado significativamente a través de mutaciones en el curso de la evolución, las células de las nuevas especies siguen construyendo los mismos tipos de máquinas, usando para ello los componentes alterados». Las graduales sustituciones de letras en el ADN. que van alterando poco a poco la secuencia de aminoácidos de una proteína —y que siempre han hecho las delicias de los darwinistas ortodoxos— parecen ahora menos relevantes que nunca para la generación de novedad evolutiva. En el seno de una máquina multiproteica, las lentas variaciones neodarwinianas de una proteína no tienen más remedio que venir compensadas por alteraciones en la proteína de al lado, de modo que la interacción entre las dos proteínas se mantenga y la máquina no se deshaga en pedazos. Si todas las células eucariotas están basadas en máquinas multiproteicas complejas, ajustadas y engrasadas, y si esas máquinas son en gran medida las mismas en todas las especies animales, lo más probable es que la materia prima de la innovación evolutiva no sea el gen, en su constante fluir acumulativo de cambios de aminoácidos, sino la máquina en su conjunto, que puede ser reclutada como tal en un nuevo lugar, tiempo o situación, tal vez mediante la mera sustitución de uno de sus componentes.
Si la teoría de Margulis revelaba un punto débil con el problema del splicing, el descubrimiento de que la práctica totalidad de la célula es una macrofactoría compuesta de máquinas complejas y exquisitamente imbricadas multiplica el tamaño de ese punto débil. En concreto, lo multiplica por 232. ¿Fueron un centenar de esas máquinas aportadas a la simbiosis por la arquea, y otro centenar por la bacteria? ¿O cada una aportó unos cuantos componentes a cada máquina? ¿Tienen los procariotas máquinas de este tipo, o versiones más simples? Demasiadas preguntas otra vez.
Bien, dije que iba a mostrar que la teoría de Margulis era correcta, pero incompleta, y eso es lo que he hecho. Sigamos.


